
I “Nuovi Approcci Metodologici” in inglese “New Approach Methodologies” (NAMs) sono definiti come quelle tecnologie, metodologie, approcci avanzati che possono essere utilizzati per ottenere informazioni sul rischio associato alle sostanze chimiche e che non prevedono l’uso di animali interi. La loro applicazione però si estende anche oltre lo studio degli effetti delle sostanze chimiche e spazia dalla ricerca biomedica alla didattica.
I notevoli progressi nelle biotecnologie, nella microingegneria, microfluidica, informatica, nella potenza di calcolo e nella rispettiva cooperazione multidisciplinare, hanno consentito lo sviluppo di Nuovi Approcci Metodologici (NAMs), inaccessibili solo fino a pochi anni fa (1). I NAMs permettono di studiare le condizioni e le malattie umane in un’ottica human-based, cioè incentrata sulla biologia umana ed in condizioni fisiologicamente rilevanti, svolgendo un ruolo importante nella ricerca tossicologica e biomedica (2, 3).

I NAMs si sono già dimostrati migliori dei modelli animali nel predire le risposte umane (4-9,14). Qualora promossi ed applicati con un approccio integrato, i NAMs human-based permettono da una parte lo sviluppo di farmaci e vaccini più sicuri ed efficaci per la specie umana e dall’altra il superamento della sperimentazione animale.
Il database NAT (Non-Animal Technologies) contiene informazioni sulle moderne tecnologie non animali provenienti da varie aree della biomedicina e delle scienze della vita, basate su studi scientifici e pubblicazioni.
ATTENZIONE, la maggior parte dei NAMs disponibili e già utilizzati con successo nell’ambito della ricerca biomedica accademica e di base NON sono stati ancora convalidati ai fini regolatori, non possono essere cioè attualmente utilizzati al posto degli animali per fini regolatori.
Segue un elenco non esaustivo dei principali NAMs applicabili alla ricerca biomedica;
Tecniche di imaging / scansione avanzate
Esiste una vasta gamma di tecnologie di scansione che possono rivelare i processi in vivo, in modo non invasivo, negli esseri umani. Le immagini prodotte ad elevatissima risoluzione sono particolarmente utili nello studio del cervello, delle malattie neurodegenerative come il morbo di Parkinson e l’Alzheimer. Alcuni esempi sono la Risonanza Magnetica (MRI) la Risonanza magnetica funzionale, (fMRI), la tomografia assiale computerizzata (TAC) con ricostruzione tridimensionale, la tomografia ed emissione di positroni (PET). Queste tecnologie, che possono essere combinate tra loro o con altre tecniche, permettono di studiare in modo non invasivo non solo le strutture anatomiche ma anche la composizione biochimica ed il metabolismo dei vari organi. Esempi di applicazione:
- Studio del cervello: Guarda video
- Malattia di Alzheimer: studio dell’associazione tra dieta, sindrome metabolica e Alzheimer (10)
- Biologia del cancro (11)
Tessuti umani ricostituiti in vitro / Ingegneria tissutale
I tessuti umani possono essere anche ricostituiti in vitro a partire dai singoli tipi cellulari, mirando a riprodurre l’architettura originaria del tessuto in vivo. Grazie ai recenti sviluppi delle nelle tecniche di stampa 3D, è possibile la ricostituzione di diversi tessuti umani con un livello di precisione elevato, per la ricerca scientifica. Esempi di tessuti ricostituiti in 3D sono l’epidermide, l’epitelio corneale, orale e gengivale, l’epitelio vaginale e delle vie respiratorie. L’ingegneria tissutale è la scienza che studia la possibilità di rigenerare organi e tessuti del corpo umano. Sebbene in origine sia nata a scopi terapeutici (rigenerazione/sostituzione di tessuti o organi danneggiati), negli ultimi anni sta assumendo una grande importanza nello sviluppo di modelli sperimentali human-based per la ricerca. Sono stati già creati in laboratorio diversi tessuti o sistemi quali ad esempio il linfonodo umano o l’articolazione.
Esempi di applicazione:
- Modelli di malattie respiratorie, inclusi tumori, infezioni, allergie: http://www.epithelix.com/products/
Tessuti ed organi umani
Le materie prime di origine umana possono essere ottenute e utilizzate in vari modi, dalla donazione post mortem, a quella in vivo (es. DNA, sangue). Fettine intatte di tessuto umano, ottenute eticamente da pazienti sottoposti ad interventi chirurgici o biopsie, possono essere mantenute in laboratorio in modo che preservino la loro funzione. Le biopsie tumorali, ad esempio, possono essere utilizzate per vedere se un farmaco si è legato al target molecolare previsto. Il confronto tra organi donati, sani e malati può fornire importanti informazioni sui processi patologici. Anche le cellule staminali di origine umana hanno un’enorme utilità nella ricerca.
Cellule staminali pluripotenti indotte (IPS)
Le cellule staminali pluripotenti sono cellule “immature” in grado di differenziarsi nei diversi tipi cellulari che compongono l’organismo. Visto che normalmente questo tipo di cellule si trova all’interno dell’embrione ai primi stadi di sviluppo, fino a non molti anni fa, per l’ottenimento di cellule staminali pluripotenti umane sarebbe stato necessario distruggere degli embrioni umani; ciò che ne limita molto l’utilizzo per questioni etiche. Grazie alle scoperte del gruppo del Prof. Yamanaka dell’Università di Kyoto (Giappone, 2007), oggi le cellule staminali pluripotenti possono essere ricavate in modo non invasivo direttamente a partire dalle cellule del paziente, ad esempio quelle del derma della pelle, ed indotte a differenziarsi in vitro nei vari tipi cellulari del corpo umano (come ad esempio cellule neuronali, pancreatiche, cardiache ed epatiche), senza dover ricorrere agli embrioni. Tali cellule, prendono il nome di “cellule staminali pluripotenti indotte” (IPS, acronimo derivante dall’inglese “Induced Pluripotent Stem cells”). Spesso la ricerca è limitata dalla difficoltà di accesso a pazienti e dalla ridotta disponibilità di tessuti freschi. Le IPS umane possono essere impiegate quali fonti per la produzione indefinita di tutti quei tipi cellulari altrimenti inaccessibili o a limitata sopravvivenza. Inoltre con le IPS è possibile fare qualcosa che non è mai stato possibile fare nella storia della ricerca biomedica: ottenere virtualmente tutti i tipi cellulari dallo stesso paziente. Dato che le IPS portano gli stessi geni e mutazioni dei pazienti dai quali provengono, i ricercatori possono usarle per ricreare le malattie in laboratorio e studiare come la genetica di un paziente e le condizioni ambientali contribuiscano alla sua malattia, aprendo le porte ad una nuova era nella medicina personalizzata.
Organoidi umani
Un organoide è un cluster cellulare in vitro in 3D, una versione semplificata e su scala ridotta di un organo, che ne ricapitola l’architettura e la funzione negli aspetti più salienti. Un organoide ha una complessa struttura multicellulare dove le cellule, quando sottoposte ad adeguati stimoli biochimici in vitro, si differenziano, si auto–assemblano e si auto–organizzano in tessuti, ricapitolando ciò che accade nell’embrione nelle prime settimane di sviluppo. Essi possono trovare impiego in:
– studio del meccanismo regolatorio dell’organogenesi;
– modellizzazione dei disturbi umani:
a) malattie infettive;
b) malattie ereditarie;
c) neoplasie.
– test di tossicità ed efficacia farmacologica;
– medicina personalizzata;
Sono stati sviluppati diversi tipi di organoidi, tra cui quelli cerebrali. Gli organoidi possono essere prodotti a partire dalle cellule staminali pluripotenti indotte (iPSCs), ricavate dai pazienti.
Bioreattori fluidici multicompartimentali modulari
Sono sistemi in vitro avanzati che grazie alla presenza di un circuito fluidico e di una pompa peristaltica permettono l’interazione dinamica tra colture e co–colture cellulari, alloggiate in camere o moduli, collegati tra loro. Ogni modulo rappresenta un organo del corpo umano e connettendo i moduli tra loro in serie o in parallelo attraverso un circuito fluidico che mima il flusso sanguigno è possibile modellare l’interazione tra organi e sistemi similmente a quanto accade in vivo.
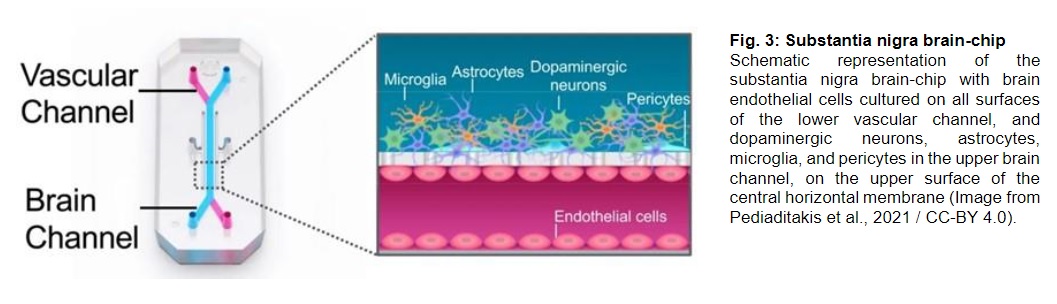
Sistemi micro-fisiologici (sistemi multi-organo su chip)
Il concetto di sistema microfisiologico (organi su chip, organoidi su chip, corpo umano su chip) si è recentemente evoluto ed è descritto come un sistema di colture cellulari, ognuna a rappresentare un tessuto o un organo che interagiscono tra loro a diversi livelli su un microchip, attraverso un circuito microfluidico, in condizioni strettamente controllate. Grazie ai sensori integrati, è possibile il monitoraggio in tempo reale delle risposte cellulari agli stimoli meccanici o chimici con un controllo più preciso dell’ambiente cellulare rispetto ai metodi convenzionali. Il sistema permette di mimare le interazioni tra cellule, tessuti (organ on a chip) ed addirittura organi e sistemi diversi (human on a chip), e di fornire gli stimoli meccanici, strutturali e biochimici adeguati, riproducendo a tutti i livelli ciò che avviene in vivo. L’utilizzo di cellule prelevate dal singolo paziente per l’assemblaggio di human on a chip personalizzati rappresenta una delle più grandi promesse per il prossimo futuro della medicina.
Scienze “omiche”
Le scienze “omiche” si occupano dello studio di pool di molecole biologiche (es.acidi nucleici, proteine, enzimi) in determinati campioni biologici (per esempio, siero, urine, liquor, saliva, tessuti). Esse analizzano, nel loro insieme: (a) i geni del DNA (genomica) e le loro funzioni (genomica funzionale); (b) i trascritti del DNA, cioè l’RNA (trascrittomica); (c) le proteine (proteomica); (d) i metaboliti all’interno di un organismo (metabolomica). Studiano anche le interazioni tra queste molecole (interattomica) e tra queste molecole ed i fattori ambientali (esposomica), i nutrienti (nutrigenomica), i fattori epigenetici (epigenomica), ecc. Lo scopo di tale approccio olistico è quello di poter comprendere operando con approcci integrativi, principi operativi di livello più elevato, che nel complesso definiscono la biologia dei sistemi. Ciò al fine di potere rispondere a domande biologiche gerarchicamente più complicate (per esempio, patogenesi, storia naturale o successo terapeutico e prognosi di una malattia). Si avvalgono dell’impiego di tecniche di analisi genetica comparativa (array-CGH) o di variazioni del numero di copie di determinati tratti del DNA (CNV) o di sequenziamento del DNA, citometria, tecniche di analisi a singola cellula, spettrometria di massa e metodi computazionali che analizzano dati di decine, centinaia o migliaia di molecole/campioni.
Metodi computazionali e “read-across“
I sistemi umani, dai singoli organi a tutto il corpo, possono essere simulati utilizzando programmi informatici altamente sofisticati. Questi vengono creati utilizzando i dati ottenuti dalle persone. Simulazioni al computer sono state sviluppate, ad esempio, per prevedere il comportamento di un farmaco nel sistema digestivo. È probabile che queste simulazioni prevedano tali effetti nell’uomo in modo più accurato rispetto ai modelli animali e in un modo molto più efficiente.
Il read-across si avvale di informazioni pertinenti relative a sostanze analoghe (“di base”) per prevedere le proprietà delle sostanze “bersaglio”. Se il read-across viene applicato correttamente, è possibile ridurre i test sperimentali in quanto non occorre sottoporre a prova ogni sostanza bersaglio.
Microdosing
Una delle caratteristiche più importanti di un candidato farmaco è il suo profilo farmacocinetico, ovvero il modo in cui il farmaco viene assorbito, distribuito, metabolizzato, escreto dall’organismo umano. Purtroppo i dati farmacocinetici forniti dai modelli preclinici tradizionali, che siano in vitro o in vivo (modelli animali) spesso non sono affidabili, in quanto non rilevanti per la biologia umana. Non a caso, la maggiore causa di fallimento nello sviluppo dei farmaci viene attribuita all’incapacità di ottenere precocemente dati farmacocinetici rilevanti per l’uomo. Concentrazioni troppo basse di farmaco a livello dell’organo target, per un tempo troppo breve, possono essere causa di inefficacia mentre concentrazioni troppo elevate, per un tempo troppo lungo, potrebbero indurre effetti tossici.
Un approccio sperimentale utile a superare questi problemi è il microdosing, che consiste nella somministrazione a volontari sani di dosi estremamente piccole, non attive farmacologicamente, di un determinato farmaco per stabilirne il profilo farmacocinetico nell’uomo. Il microdosing si basa su tecnologie analitiche ultrasensibili capaci di misurare quantità e concentrazioni infinitesimali (dell’ordine del picogrammo o del femtogrammo, cioè un billionesimo di grammo e un un milionesimo di miliardesimo di grammo!) di farmaco e di metaboliti. Le tecnologie più utilizzate a questo scopo sono la cromatografia liquida in associazione con la spettrometria di massa in tandem, la spettrometria di massa ultrasensibile con acceleratore e la tomografia ad emissione di positroni (PET).
Approccio integrato
Queste metodologie ed approcci, integrati tra loro e con gli studi interventistici ed osservazionali sugli esseri umani (pazienti e sani), costituiscono degli strumenti di fondamentale importanza per l’avanzamento della ricerca biomedica e tossicologica.
Bibliografia
1. Krewski D, Acosta D, Jr., Andersen M, Anderson H, Bailar JC, 3rd, Boekelheide K, et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of toxicology and environmental health Part B, Critical reviews. 2010;13(2-4):51-138.
2. Langley GR, Adcock IM, Busquet F, Crofton KM, Csernok E, Giese C, et al. Towards a 21st-century roadmap for biomedical research and drug discovery: consensus report and recommendations. Drug Discov Today. 2017;22(2):327-39.
3. Busquet F, Hartung T, Pallocca G, Rovida C, Leist M. Harnessing the power of novel animal-free test methods for the development of COVID-19 drugs and vaccines. Archives of toxicology. 2020.
4. Barrile R, van der Meer AD, Park H, Fraser JP, Simic D, Teng F, et al. Organ-on-Chip Recapitulates Thrombosis Induced by an anti-CD154 Monoclonal Antibody: Translational Potential of Advanced Microengineered Systems. 2018;104(6):1240-8.
5. Hartung T. AI more accurate than animal testing for spotting toxic chemicals 2019 [Available from: https://theconversation.com/ai-more-accurate-than-animal-testing-for-spotting-toxic-chemicals-99708.
6. Goyal G, Long J, Ingber DE. Microenginered human lymphoid tissue on chip. Cancer Immunology Research. 2018;6(9 Supplement):A76.
7. Ahmed S, Bibby L, Dickinson A. Predicting adverse immune reactions to biopharmaceuticals using a human in-vitro skin explant test: a promising tool for biopharmaceutical R&D development. 2017.
8. Ahmed SS, Whritenour J, Ahmed MM, Bibby L, Darby L, Wang XN, et al. Evaluation of a human in vitro skin test for predicting drug hypersensitivity reactions. Toxicology and applied pharmacology. 2019;369:39-48.
9. Passini E, Britton OJ, Lu HR, Rohrbacher J, Hermans AN, Gallacher DJ, et al. Human In Silico Drug Trials Demonstrate Higher Accuracy than Animal Models in Predicting Clinical Pro-Arrhythmic Cardiotoxicity. 2017;8(668).
10. Pistollato F, Cano SS, Elio I, Vergara MM, Giampieri F, Battino M. The Use of Neuroimaging to Assess Associations Among Diet, Nutrients, Metabolic Syndrome, and Alzheimer’s Disease. J Alzheimers Dis. 2015;48(2):303-18.
11. García-Figueiras R, Baleato-González S, Padhani AR, Luna-Alcalá A, Vallejo-Casas JA, Sala E, et al. How clinical imaging can assess cancer biology. Insights into Imaging. 2019;10(1):28.
12. Madden LR, Nguyen TV, Garcia-Mojica S, Shah V, Le AV, Peier A, et al. Bioprinted 3D Primary Human Intestinal Tissues Model Aspects of Native Physiology and ADME/Tox Functions. iScience. 2018;2:156-67.
13. Kraus T, Lubitz A, Schließer U, Giese C, Reuschel J, Brecht R, et al. Evaluation of a 3D Human Artificial Lymph Node as Test Model for the Assessment of Immunogenicity of Protein Aggregates. Journal of pharmaceutical sciences. 2019;108(7):2358-66.
14. Ewart, L., Apostolou, A., Briggs, S.A. et al. Performance assessment and economic analysis of a human Liver-Chip for predictive toxicology. Commun Med 2, 154 (2022).



1 Trackback / Pingback
I commenti sono bloccati.