Come i NAM contribuiscono all’avanzamento della ricerca su Covid-19: qualche esempio
La ricerca attraverso Nuovi Approcci Metodologici (NAM) in vitro ed in silico focalizzati sulla biologia umana (human-based) e lo studio dei pazienti ha portato e sta portando a nuove ed interessanti scoperte nel campo della ricerca riguardante il nuovo Coronavirus (SARS-CoV-2 ovvero coronavirus 2 da sindrome respiratoria acuta grave) e la malattia causata dallo stesso (malattia da coronavirus 2019 o COVID-19). Ad esempio i ricercatori dell’ Hubrecht Institute in Utrecht, dell’Erasmus MC University di Rotterdam dell’Università di Mastricht in Olanda, stanno utilizzando delle colture tridimensionali (3D) ingegnerizzate di cellule intestinali derivate dai pazienti (i così detti organoidi) e tecniche avanzate di sequenziamento dell’RNA per studiare come SARS-CoV-2 interagisce con il sistema gastrointestinale umano, sulla base di osservazioni cliniche secondo le quali il virus causerebbe in alcuni casi anche sintomi gastrointestinali. Grazie a questo modello gli scienziati hanno scoperto che SARS-CoV-2 è in grado di infettare le cellule intestinali e di riprodursi nell’intestino, sfruttando un recettore specifico, presente in gran quantità sulle cellule intestinali umane. I ricercatori stanno attualmente esplorando le differenze tra infezioni polmonari e intestinali confrontando gli organoidi polmonari e intestinali infettati da SARS-CoV-2 (1, 2). Il prossimo passo sarà quello di integrare nel modello il microbiota intestinale umano (i batteri simbionti residenti nell’intestino) e cellule del sistema immunitario per studiare le complesse relazioni tra il virus, i batteri intestinali ed il sistema immunitario. Gli organoidi umani hanno permesso anche di ottenere importanti informazioni sulla relazione tra malattie infiammatorie intestinali, genetica e meccanismi con cui Covid19 entra nelle cellule intestinali umane (3).
Colture 3D di cellule staminali polmonari umane hanno consentito di modellare l’infezione da SARS-CoV-2, di identificare le “firme molecolari” della risposta immunitaria, e di fare luce sui meccanismi sottostanti la malattia (4).
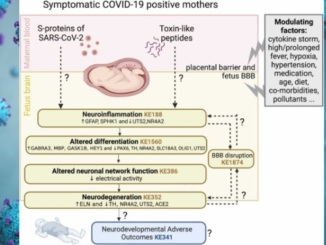
Utilizzando tessuti post-mortem provenienti da pazienti affetti da Covid-19, un team di ricercatori della Charité – Universitätsmedizin di Berlino ha scoperto il meccanismo attraverso cui il virus può raggiungere il cervello e come il sistema immunitario risponde all’invasione. I risultati, che dimostrano che il virus penetra nel cervello attraverso le cellule nervose presenti nella mucosa olfattiva, sono stati recentemente pubblicati su Nature Neuroscience (5).
I ricercatori del CAAT (Center for Alternatives to Animal Testing) hanno scoperto per la prima volta che SARS-CoV-2 può infettare il cervello e replicarsi, grazie agli organoidi cerebrali umani (mini-brains) (6).
Al Wyss Institute dell’Università di Harvard (USA) si lavora su approcci multidisciplinari, integrando tecnologie di organi umani su chip, intelligenza artificiale e machine learning, per lo studio delle modalità di sviluppo dell’infezione, della diagnosi e terapia per COVID-19. L’equipe del Prof. Ingber, direttore dello stesso centro, sta lavorando su sistemi microfluidici (vie respiratorie e polmone umano su chip) per studiare l’entrata e la replicazione del virus nelle cellule, la risposta immunitaria e per testare l’efficacia dei farmaci capaci di inibire il virus. Il sistema si è dimostrato in grado di replicare l’effetto dei farmaci antivirali riscontrato nei pazienti .
L’intelligenza artificiale è un altro approccio che si può utilizzare per integrare ed analizzare grandi quantità di dati derivanti dai pazienti, al fine di comprendere meglio le modalità di diffusione del virus, migliorare l’accuratezza e la velocità della diagnosi, e sviluppare nuovi approcci terapeutici, identificando gli individui più a rischio a seconda delle caratteristiche genetiche e dello stile di vita. Tecniche avanzate di “machine learning” sono state impiegate con successo in diverse situazioni tra cui la classificazione dei diversi ceppi del virus, la previsione della sopravvivenza di pazienti con forme severe di malattia, e la scoperta di nuovi candidati farmaci contro COVID-19 (7).
Anche in Italia, al centro di ricerca Piaggio di Pisa, il gruppo di ricerca guidato dal bioingegnere Ahluwalia sta mettendo a punto un modello innovativo di polmone 3D che replica i movimenti respiratori e che integrato con cellule di pazienti o guariti, permette di studiare l’interazione tra gli alveoli polmonari e l’aerosol contenente nanoparticelle o virus.
Grazie alle ultime frontiere delle tecnologie così dette “omiche” è possibile effettuare saggi rapidi analizzando grandi quantità di dati in poco tempo. Ad esempio attraverso analisi multi-parametriche a singola cellula è possibile esaminare il profilo immunitario di grandi campioni di popolazione. Ciò permette di studiare la risposta immunitaria umana nella sua unica complessità (4, 8, 9). Studi su larga scala dei genomi di cellule polmonari umane hanno permesso di individuare i geni ed i percorsi biologici potenzialmente implicati nell’infezione delle cellule polmonari, fornendo informazioni vitali sui meccanismi di infezione e di malattia e quindi sui potenziali target terapeutici. Grazie ad avanzate tecniche di editing genomico applicate alle cellule umane in vitro è stato possibile determinare quali sono i geni che proteggono dall’infezione e caratterizzarne le funzioni biologiche (10).
Le tecniche in vitro ed in silico, soprattutto se utilizzate in modo integrato e combinate con gli studi clinici ed epidemiologici su larga scala possono fornire risultati più affidabili e rilevanti per i pazienti, evitando i pericoli derivanti da un sovra-affidamento ai modelli animali, che spesso non sono in grado di riflettere la complessità delle condizioni umane. In particolare per quanto riguarda SARS-CoV-2, i modelli animali, anche se infettati dal virus, non presentano gli stessi sintomi riscontrati nei pazienti, ed il loro sistema immunitario risponde in maniera differente da quello umano, rendendo molto difficili gli studi finalizzati alla scoperta di nuovi farmaci e vaccini efficaci. Ricordiamo che l’85-95% dei farmaci che danno ottimi risultati sugli animali si rivelano poi inefficaci se non addirittura pericolosi per i pazienti (11-15).
Non a caso la comunità scientifica riconosce sempre di più la necessità di focalizzare la ricerca sulla biologia umana (16, 17), proprio perché i modelli animali, per quanto possano essere migliorati o “umanizzati”, non sono in grado di riflettere le risposte umane, mentre con i loro elevati costi sottraggono ingenti risorse alla ricerca.
Un articolo recentemente pubblicato sulla rivista Archives of Toxicology da 5 noti ricercatori del CAAT illustra una panoramica di approcci innovativi che potrebbero essere impiegati per la ricerca su SARS-CoV-2. Questi NAM human-based sono già disponibili e come spiegato sopra hanno già iniziato a dare preziosi contributi alla ricerca su COVID-19 nonostante si tratti di approcci e tecnologie emergenti, con un grande potenziale per il futuro. Tuttavia purtroppo il 95% delle risorse per la ricerca preclinica – osservano gli autori – è diretto verso la sperimentazione animale e generazione di nuovi modelli animali, che non essendo in grado di riflettere accuratamente la malattia umana, potrebbero fuorviare i ricercatori fino a mettere addirittura a rischio la sicurezza dei volontari umani durante le fasi cliniche di sperimentazione dei farmaci e dei vaccini (18).
Quanto più ci impegneremo a promuovere i nuovi approcci incentrati sulla biologia umana sia a livello economico che formativo, tanto prima questi diverranno i metodi di riferimento per la ricerca biomedica e tossicologica, con notevole beneficio per i pazienti in attesa di cure, per la collettività, per la ricerca e per gli animali.
Bibliografia
1. Lamers MM, Beumer J, van der Vaart J, Knoops K, Puschhof J, Breugem TI, et al. SARS-CoV-2 productively infects human gut enterocytes. Science (New York, NY). 2020.
2. Mini-organs push along Covid-19 and other virus research [Available from: https://knowablemagazine.org/article/health-disease/2020/organoids-for-covid-19-research.
3. Verstockt B, Verstockt S, Abdu Rahiman S, Ke B-j, Arnauts K, Cleynen I, et al. Intestinal Receptor of SARS-CoV-2 in Inflamed IBD Tissue Seems Downregulated by HNF4A in Ileum and Upregulated by Interferon Regulating Factors in Colon. Journal of Crohn’s and Colitis. 2020.
4. Youk J, Kim T, Evans KV, Jeong Y-I, Hur Y, Hong SP, et al. Three-Dimensional Human Alveolar Stem Cell Culture Models Reveal Infection Response to SARS-CoV-2. Cell stem cell. 2020;27(6):905-19.e10.
5. Meinhardt J, Radke J, Dittmayer C, Franz J, Thomas C, Mothes R, et al. Olfactory transmucosal SARS-CoV-2 invasion as a port of central nervous system entry in individuals with COVID-19. Nature Neuroscience. 2020.
6. Bullen CK, Hogberg HT, Bahadirli-Talbott A, Bishai WR, Hartung T, Keuthan C, et al. Infectability of human BrainSphere neurons suggests neurotropism of SARS-CoV-2. Altex. 2020;37(4):665-71.
7. Alimadadi A, Aryal S, Manandhar I, Munroe PB, Joe B, Cheng X. Artificial intelligence and machine learning to fight COVID-19. Physiol Genomics. 2020;52(4):200-2.
8. Zhang J-Y, Wang X-M, Xing X, Xu Z, Zhang C, Song J-W, et al. Single-cell landscape of immunological responses in patients with COVID-19. Nature Immunology. 2020;21(9):1107-18.
9. Liao M, Liu Y, Yuan J, Wen Y, Xu G, Zhao J, et al. The landscape of lung bronchoalveolar immune cells in COVID-19 revealed by single-cell RNA sequencing. medRxiv. 2020:2020.02.23.20026690.
10. Daniloski Z, Jordan TX, Wessels H-H, Hoagland DA, Kasela S, Legut M, et al. Identification of Required Host Factors for SARS-CoV-2 Infection in Human Cells. Cell.
11. Hartung T. Look back in anger – what clinical studies tell us about preclinical work. Altex. 2013;30(3):275-91.
12. NCATS-National-Insitute-of-Health-NIH. Transforming Translational Science 2018 [Available from: https://ncats.nih.gov/files/NCATS-factsheet.pdf.
13. DiMasi JA, Feldman L, Seckler A, Wilson A. Trends in risks associated with new drug development: success rates for investigational drugs. Clinical pharmacology and therapeutics. 2010;87(3):272-7.
14. Hunig T. The storm has cleared: lessons from the CD28 superagonist TGN1412 trial. Nature reviews Immunology. 2012;12(5):317-8.
15. Attarwala H. TGN1412: From Discovery to Disaster. Journal of young pharmacists : JYP. 2010;2(3):332-6.
16. Archibald K, Tsaioun K, Kenna JG, Pound P. Better science for safer medicines: the human imperative. Journal of the Royal Society of Medicine. 2018:141076818812783.
17. Herrmann K, Pistollato F, Stephens ML. Beyond the 3Rs: Expanding the use of human-relevant replacement methods in biomedical research. Altex. 2019;36(3):343-52.
18. Busquet F, Hartung T, Pallocca G, Rovida C, Leist M. Harnessing the power of novel animal-free test methods for the development of COVID-19 drugs and vaccines. Archives of toxicology. 2020.